فهرست مطالب:
- مرحله 1: استفاده از جعبه مغز
- مرحله 2: مقدمه: تجزیه و تحلیل مقاطع
- مرحله 3: تنظیم یک تابع: LevelCurveTracings.m
- مرحله 4: توسعه الگوریتم محدود: یافتن محدوده
- مرحله 5: توسعه الگوریتم محدود: ایجاد آرایه باند بیرونی
- مرحله 6: توسعه الگوریتم محدود: کار با مرکز نقطه
- مرحله 7: توسعه الگوریتم محدود: هنگامی که مرکز مرکزی متمرکز نیست
- مرحله 8: توسعه الگوریتم محدود: تداخل سوراخ ها
- مرحله 9: توسعه الگوریتم محدود: مکان یابی سوراخ ها ، نهایی شدن محدوده های مغز و حفره ها
- مرحله 10: ثبت اطلاعات: Function PatientFiles.m
- مرحله 11: ثبت اطلاعات در پرونده ها
- مرحله 12: ثبت داده ها: نمایش یک قطعه حجم مغز در طول زمان
- مرحله 13: بستن شکاف ها در فرعی ها: Subplotclose.m
- مرحله 14: کد اصلی: پاک کردن همه و درخواست ورود برای ورودی ها
- مرحله 15: کد اصلی: پردازش دسته ای تصاویر
- مرحله 16: کد اصلی: Padding
- مرحله 17: کد اصلی: تعیین مرزها
- مرحله 18: کد اصلی: تعیین ابعاد مناسب Z
- مرحله 19: کد اصلی: تعیین مختصات X و Y
- مرحله 20: کد اصلی: رسم یک ساختار سه بعدی ، یافتن حجم و ورود داده ها
تصویری: جعبه مغز: ردیابی حجم عصبی در طول زمان: 20 مرحله

2024 نویسنده: John Day | [email protected]. آخرین اصلاح شده: 2024-01-31 10:19
پیشروی به سوی عمر طولانی تر بشر ، بیماری هایی را پدید آورده است که تمدن های پیش از ما مشاهده نکرده اند. در میان این موارد ، آلزایمر در سال 2017 تقریباً 5.3 میلیون نفر از افراد مسن آمریکایی زندگی می کرد ، یا تقریباً 1 نفر از هر 10 سالمند آمریکایی (https://www.alz.org/facts/) و تعداد بی شماری دیگر مبتلا به زوال عقل بودند. این کد برای کمک به مبارزه برای درک آنچه رنج می برد بزرگان ما ، محققان آینده و مشتاق کنجکاو را قادر می سازد تا با گذشت زمان حجم مغز را ردیابی کنند.
مرحله 1: استفاده از جعبه مغز
برای استفاده از جعبه مغز ، افراد فقط به موارد زیر نیاز دارند:
- اسکن MRI از مغز و نام و قالب چنین پرونده هایی (همه باید تقریباً دارای ابعاد یکسان باشند)
- طول یک اسکن
- فاصله بین هر لایه (اسکن MRI)
- نام بیمار (هنگام وارد کردن ، فاصله ها را وارد نکنید و لطفاً نام و نام خانوادگی را با حروف بزرگ بنویسید ، مانند: FirstnameLastname)
از این رو ، فرد می تواند روند حجم افراد را در طول زمان پیگیری کند. بنابراین ، ارقام روندهای آلزایمر توسط این نرم افزار قابل ردیابی است. طول مورد استفاده ما در آزمایش 180 میلی متر برای طول یک اسکن و 5 میلی متر برای فاصله بین اسکن MRI بر اساس ارقام متوسط بود.
با این حال ، استفاده از جعبه مغز نیازی به این کار محدود نیست. اگر سطح مقطع یک جامد معین عکس دار باشد ، مانند یک تومور به خودی خود ، روند تغییرات حجم برای اینها را می توان در نرم افزار پیگیری کرد.
مرحله 2: مقدمه: تجزیه و تحلیل مقاطع
در ساختارهای سه بعدی ، به صفحه های دو بعدی که چنین تشکیل شده اند ، مقطع می گویند. تصور کنید که یک دسته کاغذ منشور مستطیلی را تشکیل می دهد ، سپس هر تکه کاغذ مقطع کاغذ خواهد بود. در تصور مغز ، ما از همان سیر فکری استفاده می کنیم. MRI (تصویربرداری رزونانس مغناطیسی) (به اطلاعات مربوط به MRI مراجعه کنید) مقاطع مختلف مغز را ضبط می کند و با استفاده از مرزهای تعریف شده در هر "لایه" از مغز ارائه شده ، می توانیم ساختاری برای مدل سازی و یافتن حجم مغز ایجاد کنیم. به ما ابتدا باید یک تابع ایجاد کنیم تا اطلاعاتی را در مورد چنین مرزهایی ارائه دهیم.
مرحله 3: تنظیم یک تابع: LevelCurveTracings.m
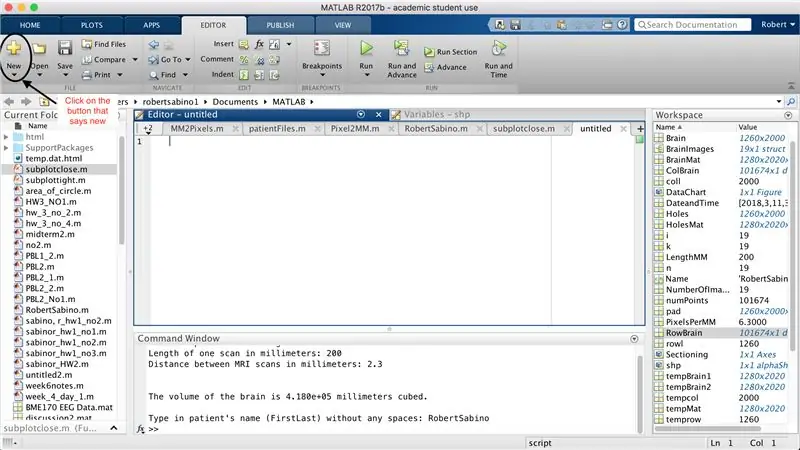
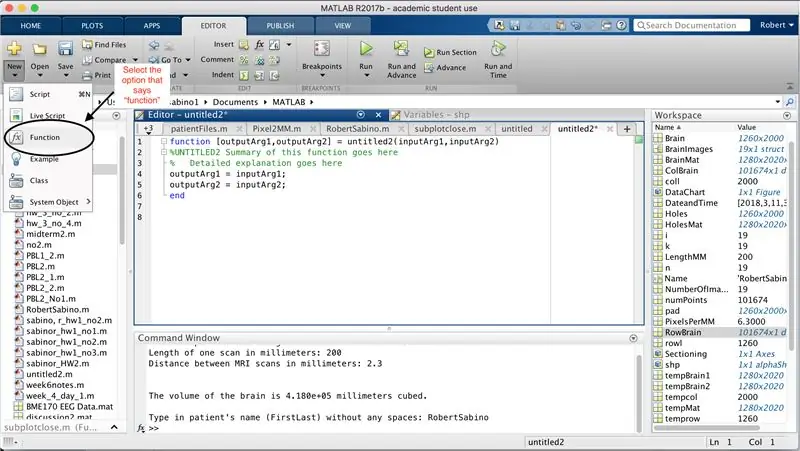
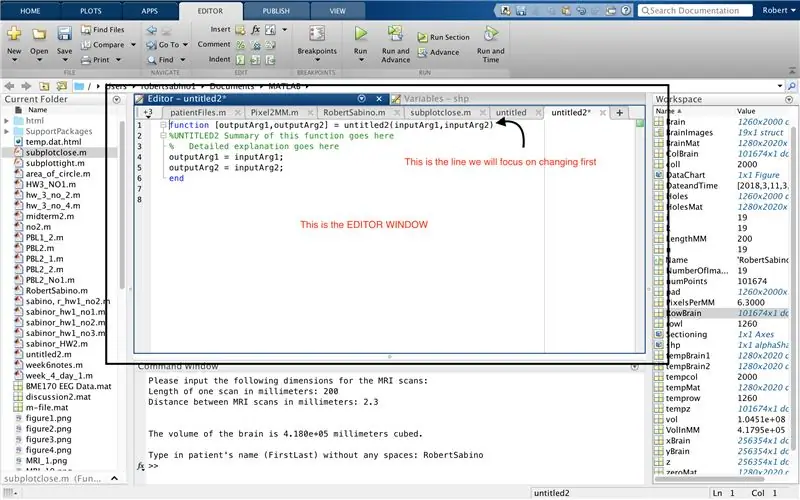

ابتدا مطمئن شوید MATLAB_R2017b رایانه شما بارگیری شده است (اینجا را بارگیری کنید) و MATLAB را باز کنید. در رابط MATLAB ، روی دکمه در گوشه سمت چپ بالای پنجره که روی آن نوشته شده "New" با علامت زرد پررنگ بعلاوه کلیک کنید ، و گزینه "function" را انتخاب کنید ، تا در پنجره ویرایشگر فضایی شبیه به آنچه در عکس سوم ما بر روی تغییر خط اول برای تنظیم عملکرد تمرکز می کنیم. در جایی که می گوید "outputArg1" ، آن را با "مغز" ، "outputArg2" برای گفتن "حفره ها" ، "بدون عنوان 2" تا "exp2" و "inputArg1" به "تصویر" جایگزین کنید و "inputArg2" را حذف کنید. اکنون شما باید با استفاده از "exp2" ، یک آرگومان "تصویر" و خروج مرزهای "مغز" و "حفره ها" از آن استفاده کنید. خط اول تابع باید شبیه خطی باشد که در تصویر چهارم نشان داده شده است. تمام کد زیر این خط اولیه را حذف کنید.
مرحله 4: توسعه الگوریتم محدود: یافتن محدوده

کد زیر را در زیر خط وارد کنید. این بخش از تابع خط به خط زیر را انجام می دهد.
- تصویر "image" را در متغیر "mri" بارگذاری کنید.
- "mri" را به یک تصویر ساخته شده از مقادیر در محدوده اعداد به یک و صفر (با نام مستعار بایناریزه) بر اساس مقدار تعیین شده آستانه تبدیل کنید. اگر مقدار پیکسل برابر یا بزرگتر از 0.1 باشد ، روی یک تنظیم می شود ، اگر نه ، مقدار آن پیکسل صفر است.
- چهار خط زیر 10 ستون و ردیف در لبه های لایه اسکن MRI را به صفر تبدیل می کند تا از خواندن مقادیر نامناسب بعنوان محدود کننده جلوگیری شود (همانطور که از آزمایش با کد آموخته شد).
- در خط آخر ، bwboundaries مرزهای تصویر دوتایی "mri" را ردیابی کرده و آن را برابر با "b" قرار می دهد ، یک آرایه با عناصری که شاخص های آنها با محدوده های تعیین شده یک برابر است.
مرحله 5: توسعه الگوریتم محدود: ایجاد آرایه باند بیرونی
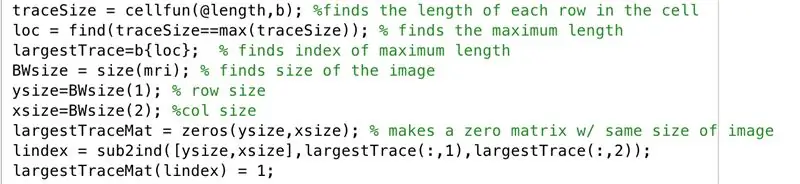
در پنجره ویرایشگر کد زیر را در تصویر دنبال کنید. این بخش از کد خط به خط زیر را انجام می دهد.
- طول هر یک از سطرهای تصویر دوتایی "b" را بیابید (cellfun طول تابع را در هر سطر اعمال می کند).
- "loc" را برای ذخیره حداکثر طول ها تنظیم کنید.
- نمایه حداکثر طول را پیدا کنید ، تنظیم شده برای ذخیره در "largeTrace".
- اندازه تصویر "mri" را که از همان اندازه "b" تشکیل شده است ، بیابید و روی "BWsize" تنظیم کنید.
- تعداد ردیف های موجود در آرایه تصویر را که روی "ysize" تنظیم شده است پیدا کنید.
- تعداد ستون های موجود در آرایه تصویر را که روی "xsize" تنظیم شده است ، بیابید.
- ایجاد آرایه "largeTraceMat" ، یک "ysize" با ماتریس "xsize" از صفر.
- شاخص معادل را از مقادیر ثبت شده پیدا کنید که مربوط به جایی است که بزرگترین مقادیر x x و y در بردار "lindex" ذخیره شده است.
- در ماتریس صفرها ، "largeTraceMat" ، عناصر شاخص هایی را که مطابق با مقادیر شاخص ذخیره شده به عنوان عناصر در "lindex" هستند ، به واحدهایی تبدیل کنید.
بنابراین آرایه منطقی "largeTraceMat" دارای بزرگترین ناحیه محدود شده از سطح مقطع اسکن مغزی داده شده است که به عنوان مواردی با پس زمینه صفر ترسیم شده است.
مرحله 6: توسعه الگوریتم محدود: کار با مرکز نقطه

در مرحله بعد ، باید آزمایش کنیم که ببینیم آیا سطح مقطع شامل بیش از یک منطقه (بزرگترین) است. با آزمایش هم ترازی مرکز مرکزی بزرگترین منطقه ، می توانیم ببینیم که آیا یک ناحیه مجاور وجود دارد که مرکز مرکزی بیشتری را تولید کند یا احتمال وجود چندین منطقه.
- از "regionProps" برای پیدا کردن اطلاعات در مورد سانتریوئیدهای موجود ، برابر با آرایه ساختار "tempStruct" استفاده کنید
- آرایه فرم "centroids" با داده های مربوط به زمینه "centroid" به صورت عمودی متصل شده است
- مقادیر ستون دوم "centroids" (مختصات بعد افقی) را در نظر بگیرید
- یک فیلتر اجرا کنید تا هم ترازی مرکز را با مرکز افقی بررسی کنید
مرحله 7: توسعه الگوریتم محدود: هنگامی که مرکز مرکزی متمرکز نیست
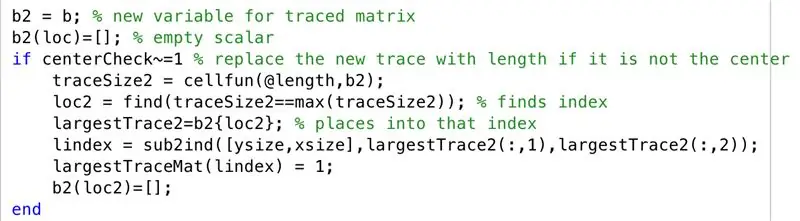
در سناریویی که مرکز اصلی بزرگترین منطقه ردیابی مرکز نباشد ، مراحل زیر را طی می کنیم. همانطور که در اسکن های MRI مشاهده کرده بودیم ، تمایل به این بود که نیمکره های مغز در مقطع به تصویر کشیده شوند ، در حالی که مجاور هم نیستند ، بنابراین ما در حال حاضر به ترسیم دومین بزرگترین اثر به همراه بزرگترین اثر در "largeTraceMat" ادامه می دهیم.
- ماتریس ردیابی شده را روی متغیر جدید "b2" تنظیم کنید
- ماتریس خالی "b2" را با مجموعه ای که توسط "loc" نمایه شده است ، اولیه کنید
- یک شرطی ایجاد کنید ، برای زمانی که یک مرکز مرکزی (مانند یک لایه چند ناحیه) متمرکز نباشد
- اندازه ردیابی جدیدی را برای هر ردیف پیدا کنید (traceSize2)
- "loc2" را برای یافتن شاخص هایی که محدوده وجود دارد تعیین کنید
- اجازه دهید سلولهای مشخص شده توسط "loc2" در "b2" برابر "largeTrace2" باشند
- تبدیل زیرنویس ها به شاخص ها ، روی "lindex"
- عناصر مربوط به "lindex" را در "largeTraceMat" به 1 تغییر دهید
- ماتریس خالی "b2" را با مجموعه ای که توسط "loc2" نمایه شده است ، اولیه کنید
مرحله 8: توسعه الگوریتم محدود: تداخل سوراخ ها

در برخورد با حفره ها ، مقادیر ذخیره شده در "b2" ساختارهای دیگری غیر از بزرگترین ردیابی را دنبال می کند ، و ترسیم آنها بر روی یک فرم پر شده از "largeTraceMat" مشخص می کند که سوراخ هایی در مناطق مغز وجود دارد.
- ایجاد آرایه "fillMat" ، که به صورت پر شده از "largeTraceMat" است
- ایجاد آرایه "interferenceMat" ، "ysize" توسط "xsize" آرایه از صفر
- ایجاد آرایه "interferenceloc" ، برای ذخیره مقادیر "b2" ، به صورت عمودی متصل شده است
- ایجاد آرایه "lindex" برای ذخیره شاخص های مربوط به "interferenceloc"
- برای شاخص های "interferenceMat" که با "lindex" مطابقت دارد ، مقدار را روی 1 تنظیم کنید ، و یک منطقه محدود متفاوت ایجاد کنید
مرحله 9: توسعه الگوریتم محدود: مکان یابی سوراخ ها ، نهایی شدن محدوده های مغز و حفره ها

- آرایه "tempMat" را برابر با "interferenceMat" به علاوه "fillMat" تنظیم کنید ، بنابراین هر مقدار را در ماتریس به یکدیگر اضافه کنید
- مجموعه آرایه "holeLoc" برابر با شاخص هایی که "interferenceMat" و "fillMat" هر دو برابر یک بودند
- راه اندازی "holeMat" به عنوان ماتریس صفر ابعاد "ysize" x "xsize"
- شاخص هایی را در "holeMat" تنظیم کنید که برابر با "holeLoc" هستند
- "مغز" را روی "largeTraceMat" تنظیم کنید
- "hole" را روی "holeMat" قرار دهید
با یافتن جایی که مقادیر ماتریس های اضافه شده برابر 2 بود ، مکانهای حفره به راحتی ایمن شده و بر روی یک ماتریس خالی ترسیم شد.
مرحله 10: ثبت اطلاعات: Function PatientFiles.m
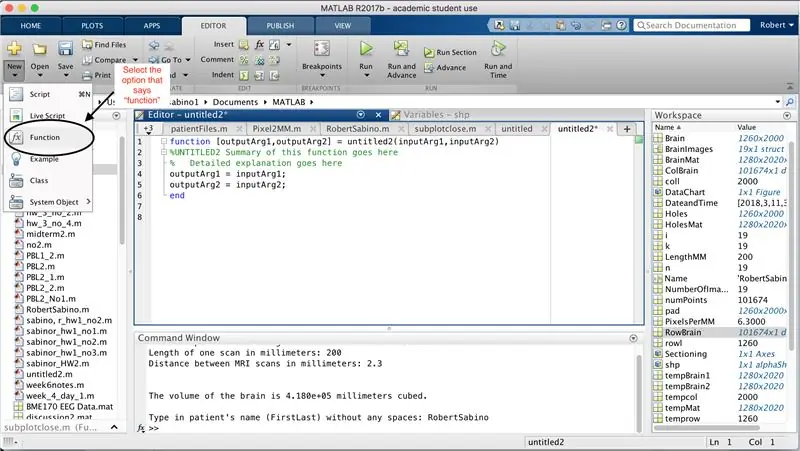
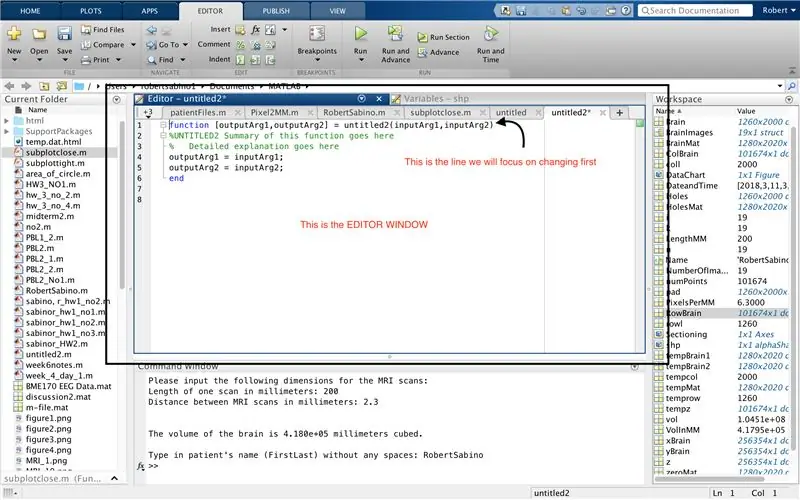

درست مانند راه اندازی آخرین تابع ، روی دکمه ای که در گوشه سمت چپ بالای پنجره قرار دارد و روی آن نوشته شده "جدید" با علامت زرد به رنگ زرد ضخیم کلیک کنید و گزینه "تابع" را انتخاب کنید تا در پنجره ویرایشگر فضایی باز شود که شبیه تصویر سوم است در خط اول ، ماتریس خروجی را حذف کرده و فقط با "خروجی" جایگزین کنید ، "untitled2" را با "pacientFiles" جایگزین کنید ، تمام آرگومان های ورودی را حذف کرده و در عوض قالب بندی مشخص شده در تصویر چهارم خط کد را دنبال کنید. خط اول این عملکرد باید با قالب بندی تصویر مطابقت داشته باشد.
مرحله 11: ثبت اطلاعات در پرونده ها
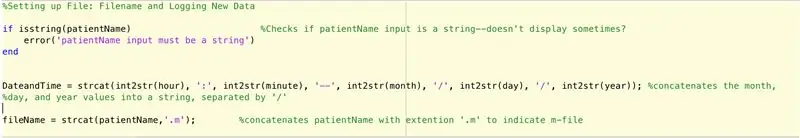

برای تنظیم یک فایل برای ثبت داده های یافت شده توسط تابع اصلی (هنوز توضیح داده نشده است) ، ما باید این مراحل را دنبال کنیم (همانطور که توسط کد خط به خط توصیه شده است).
- بررسی کنید که آیا ورودی برای pacientName یک رشته است یا خیر.
- اگر یک رشته نیست ، آن ورودی بیمار را باید یک رشته نشان دهید.
- دستور if را خاتمه دهید (از خطا جلوگیری کنید).
- یک دستور رشته "DateandTime" تنظیم کنید که قالب زیر را ارائه می دهد: ساعت: دقیقه-ماه/روز/سال.
- متغیر fileName را به صورت زیر تنظیم کنید: pacientName.m.
حال به بخش بعدی تابع: آیا فایلی با این نام از قبل وجود دارد؟
1) فرض کنید فایل با این نام در حال حاضر وجود دارد:
- فایل را اجرا کنید تا مقادیر قبلی در صف قرار گیرند
- داده های "DateandTime" تکرار فعلی را به عنوان یک سلول جدید در آرایه سلولی مقادیر x (انتهای شاخص+1) اضافه کنید
- مقدار فعلی "brainVolume" را به عنوان یک سلول جدید در آرایه سلولی مقادیر y اضافه کنید (انتهای شاخص+1)
- متغیرهای فعلی بارگذاری شده در فایل را ذخیره کنید.
2) فرض کنید پرونده ای با این نام وجود ندارد:
- ایجاد یک فایل جدید با نام ذخیره شده در متغیر "pacientName"
- داده های جاری "DateandTime" را به عنوان سلول به آرایه سلول خالی از مقادیر x اضافه کنید
- داده های فعلی "brainVolume" را به عنوان یک سلول به آرایه سلول خالی از مقادیر y اضافه کنید
- متغیرهای فعلی بارگذاری شده در فایل را ذخیره کنید.
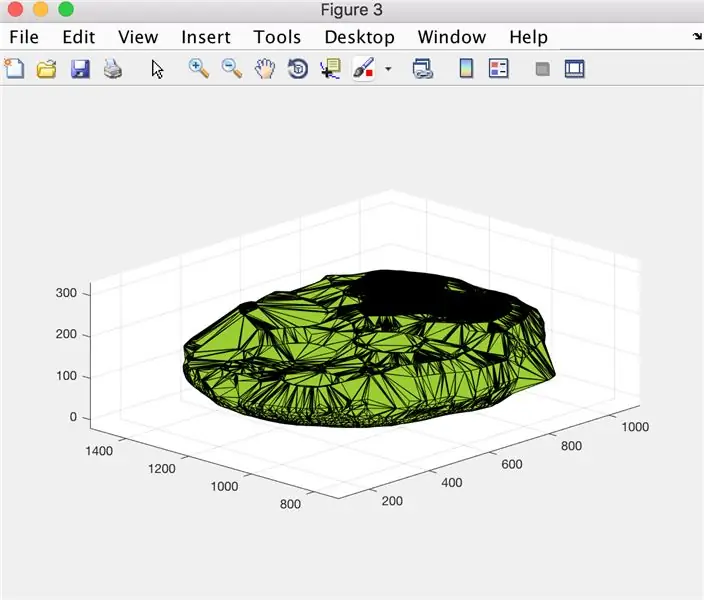
مرحله 12: ثبت داده ها: نمایش یک قطعه حجم مغز در طول زمان

- آرایه x مقادیر (xVals) را به یک آرایه دسته ای (xValsCategorical) تبدیل کنید تا ترسیم شود
- پنجره شکل 5 را ایجاد کنید
- نقاط تعیین شده توسط "xValsCategorical" و "yVals" (حاوی حجم مغز) را ترسیم کنید ، با استفاده از دایره های توخالی برای نشان دادن نقاط و اتصال آنها با خطوط تیره
- عنوان طرح را به عنوان: pacientName حجم حجم داده نامگذاری کنید
- محور x را همانطور که در تصویر نشان داده شده برچسب گذاری کنید
- مطابق تصویر ، محور y را برچسب گذاری کنید
- اجازه دهید شکل 5 برابر خروجی باشد
از این رو ، تابع بیمار که نامیده می شود ، یک فایل با داده های ویرایش شده که حجم مغز را در طول زمان ردیابی می کند ، و نمودار نمایش روند نشان می دهد.
مرحله 13: بستن شکاف ها در فرعی ها: Subplotclose.m

این تابع که از کد https://www.briandalessandro.com اقتباس شده است ، هنگامی که ارقام نمایش دهنده تصاویر MRI و لایه های مغزی ایجاد می شوند ، شکاف بین شکل های فرعی کد اصلی را برطرف می کند. تابع subplot که در subplotclose.m استفاده می شود موقعیت subplot های داده شده را طوری تنظیم می کند که دوباره از نظر ابعاد طولانی تر با یکدیگر مطابقت داشته باشند. به عنوان مثال ، اگر کد ماتریس 7 3 3 را در نظر داشته باشد ، ردیف ها به دلیل طول ابعاد طولانی تر می شوند. اگر کد ماتریس 3 7 7 داشته باشد ، ستون ها محکم می شوند و شکاف هایی در سطرها وجود دارد ، همانطور که در شکل های کد اصلی ما نشان داده شده است.
مرحله 14: کد اصلی: پاک کردن همه و درخواست ورود برای ورودی ها

برای شروع کد اصلی ، روی همان دکمه ای که در گوشه سمت چپ بالای پنجره نوشته شده است کلیک کنید و از بخشهای قبلی "Script" را به جای "Function" انتخاب کنید. کد را همانطور که در تصویر نشان داده شده است در پنجره ویرایشگر وارد کنید. خطوط کد وظایف زیر را به ترتیب انجام می دهند:
- بستن همه فایلهای باز به جز 0 ، 1 و 2.
- همه پنجره های شکل را ببندید.
- همه متغیرها را در محیط کار پاک کنید.
- پنجره Command را پاک کنید.
- نمایش در پنجره فرمان: لطفاً ابعاد زیر را برای اسکن MRI وارد کنید:
- در یک خط جدید در پنجره فرمان ، بپرسید: طول یک اسکن در میلی متر:. پاسخ داده شده توسط کاربر روی متغیر "lengthMM" تنظیم می شود.
- در خط جدید ، بپرسید: فاصله بین اسکن های MRI در میلی متر:. پاسخ داده شده توسط کاربر روی متغیر "ZStacks" تنظیم می شود.
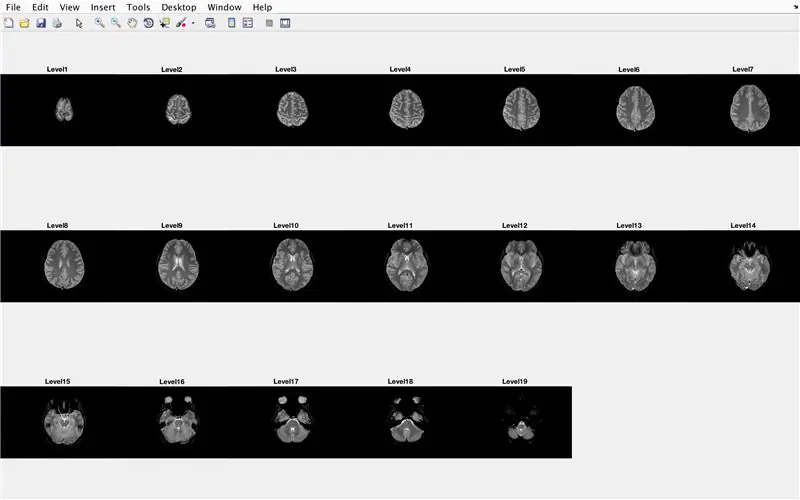
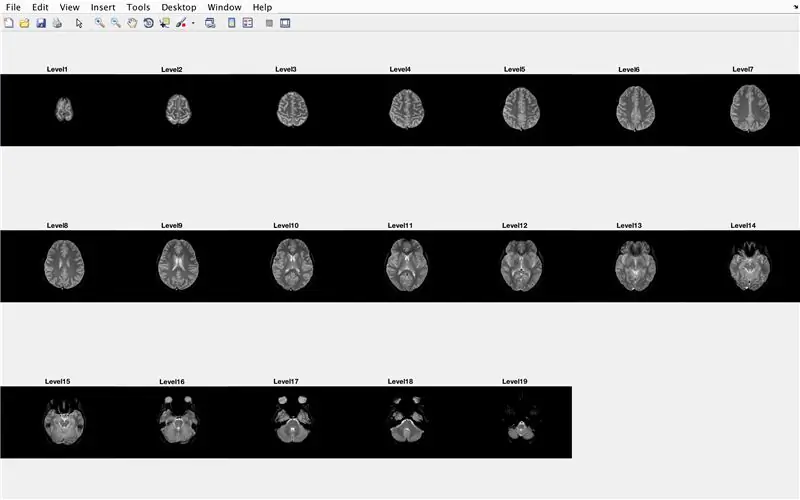
مرحله 15: کد اصلی: پردازش دسته ای تصاویر

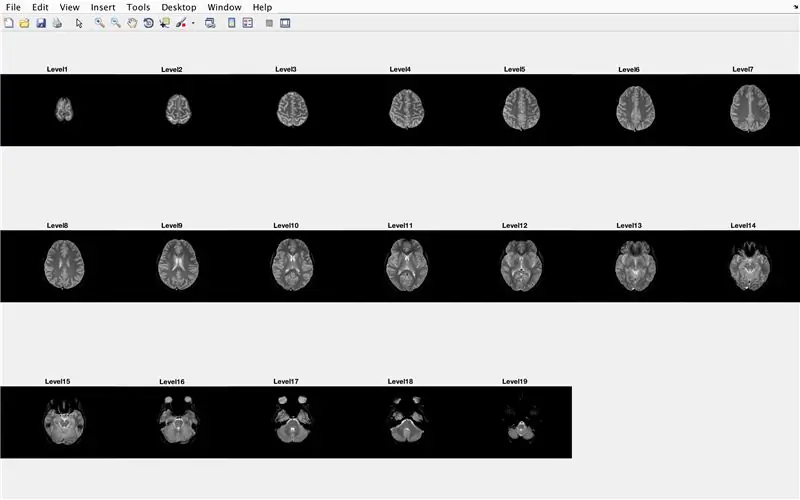
در این بخش ، کد تصاویر را بارگذاری می کند (متشکل از اسکن MRI مقاطع مختلف مغز) و نام هر فایل تصویری را در متغیر "Base" ذخیره می کند و هر یک از اسکن های MRI را نمایش می دهد. لطفاً کد موجود در تصویر را دنبال کنید ، که موارد زیر را انجام می دهد:
- ایجاد آرایه ساختار "BrainImages" که حاوی اطلاعات مربوط به تمام فایل های موجود در پوشه فعلی است که با فرمت نام MRI _ () مطابقت دارد. png
- تنظیم متغیر "NumberofImages" برابر با تعداد عناصر آرایه ساختار "BrainImages"
- پنجره شکل 1 را باز کنید
- یک حلقه for برای چرخاندن تعداد تصاویر شمارش شده در فایل تنظیم کنید
- برای هر حلقه ، "CurrentImage" نام مربوط به هر فایل MRI_i-p.webp" />
- برای ایجاد 19 تصویر که باید توسط "imshow" بارگذاری شوند ، یک زیرپلات 3 x 7 ایجاد کنید
- هر تصویر را به عنوان یک عنصر دیگر در پنجره شکل زیر نمودار نمایش دهید
- عنوان هر عنصر فرعی را Level_ بگذارید ، جایی که شماره تکرار حلقه for خالی است.
- پایان حلقه for (اجتناب از خطا)
این در پنجره شکل 1 همه اسکن های MRI را به صورت خام در پیکربندی 7 x 3 بدون هیچ شکافی در جهت x نمایش می دهد.
مرحله 16: کد اصلی: Padding

با پر کردن ، ما از وجود اختلافات جزئی در اندازه های تصویر که ممکن است برای عدم تطابق ابعاد خطا ایجاد کنند ، جلوگیری می کنیم در صورتی که یک تصویر کمی بزرگتر از دیگری باشد.
- پنجره شکل 2 را باز کنید
- بارگذاری ماتریس تصویر از MRI_1-p.webp" />
- اندازه ماتریس تصویر را بیابید و روی "OriginalXPixels" (برای تعداد سطرها) و "OriginalYPixels" (برای تعداد ستون ها) تنظیم کنید.
- ماتریس "BrainMat" را طوری تنظیم کنید که شامل همه صفرها با 20 ردیف بیشتر و 20 ستون بیشتر برای هر صفحه و 19 سطح مقطع کلی ، یک در هر صفحه باشد.
- "HolesMat" را طوری تنظیم کنید که از همان آرایه سه بعدی صفر تشکیل شده و مختصات حفره ورودی بعداً تشکیل شود.
- "zeroMat" را به اندازه پد به اضافه بیست سطر و بیست ستون ، یک آرایه دو بعدی از صفرها ایجاد کنید.
مرحله 17: کد اصلی: تعیین مرزها
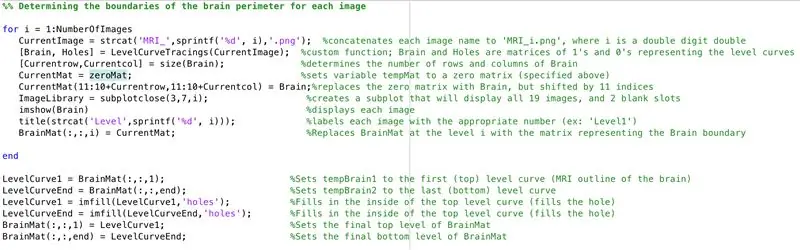
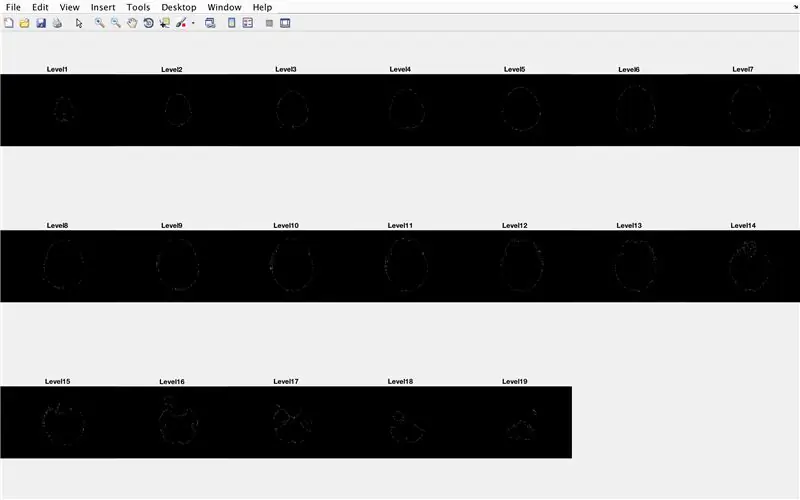
- یک حلقه for تنظیم کنید تا داده های هر تصویر بارگذاری شده قبلی را مرور کند
- در همان روش پردازش دسته ای قبلی ، "CurrentImage" فایل ها را با "MRI_i.png" بارگذاری می کند ، جایی که شماره تکرار است.
- هر تصویر را از طریق عملکرد پردازش "LevelCurveTracings2.m" که قبلاً ساخته اید اجرا کنید
- اندازه خروجی "Brain" را پیدا کنید ، تعداد ردیف ها را روی "Currentrow" و تعداد ستون ها را "Currentcolumns" تنظیم کنید.
- "CurrentMat" را روی ماتریسی از صفر با ابعاد مشخص شده توسط "Currentrow" و "Currentcolumns" تنظیم کنید
- داده های "Brain" را در "CurrentMat" قرار دهید ، با 10 ردیف در همه طرف
- برای نمایش محدوده تصاویر ، یک خرده طرح فرعی از ابعاد 3 7 7 ایجاد کنید
- عنوان هر یک از عناصر فرعی را در پنجره شکل عنوان کنید
- ایجاد ماتریس سه بعدی "BrainMat" شامل هر لایه محدود "CurrentMat"
- حلقه for را خاتمه دهید (برای جلوگیری از خطاها)
بخش زیر حفره های باقی مانده در بالا و پایین شکل سه بعدی پیشنهادی را پر می کند
- "LevelCurve1" را برابر با لایه اول "BrainMat" (پایین جامد) قرار دهید
- "LevelCurveEnd" را برابر با لایه نهایی "BrainMat" (بالای جامد) قرار دهید
- "LevelCurve1" را با یک لایه پر شده بازنویسی کنید
- "LevelCurveEnd" را با یک لایه پر شده بازنویسی کنید
- لایه پر شده را به عنوان لایه زیرین "BrainMat" تنظیم کنید
- لایه پر شده را به عنوان لایه بالای "BrainMat" تنظیم کنید
مرحله 18: کد اصلی: تعیین ابعاد مناسب Z

سه خط اول شامل تنظیم یک آرایه خالی "z" و انجام عملیات تبدیل ساده (تقسیم پیکسل ها بر طول) برای دریافت صحیح حجم در میلی متر^3 است.
- یک حلقه برای ایجاد چرخه در هر لایه ایجاد کنید
- تعداد واحدهای یک لایه معین را پیدا کنید
- مختصات z را برای مقادیر به مقادیری تبدیل کنید که به نسبت مناسب مقیاس شده اند و روی "tempz" ، بردار ستون تنظیم شده اند.
- مقدار z را برای منحنی سطح به بردار z اضافه کنید
با این کار مختصات z به درستی تنظیم می شوند.
مرحله 19: کد اصلی: تعیین مختصات X و Y

حال موقعیت x و y هر یک از نقاط موجود در مرزها را تعیین کنید.
- "xBrain" را به عنوان یک آرایه خالی اولیه کنید
- "yBrain" را به عنوان یک آرایه خالی ، اولیه کنید
- یک حلقه for برای حلقه هر تصویر بارگذاری شده تنظیم کنید
- یک ماتریس دو ستونی برای ذخیره مختصات مسطح هر نقطه در محدوده ، که توسط بردارهای ستون "RowBrain" و "ColumnBrain" نشان داده شده است ، جمع آوری کنید.
- "xBrain" را با مختصات "RowBrain" موجود پیدا کنید
- "yBrain" را با مختصات "ColumnBrain" موجود پیدا کنید
- حلقه for را خاتمه دهید (برای جلوگیری از خطا)
مرحله 20: کد اصلی: رسم یک ساختار سه بعدی ، یافتن حجم و ورود داده ها
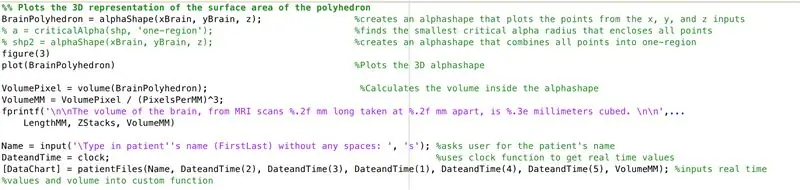

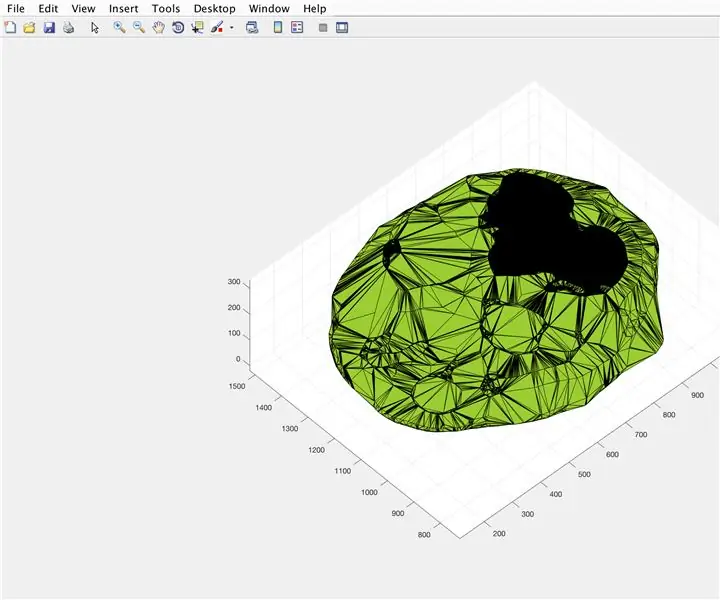
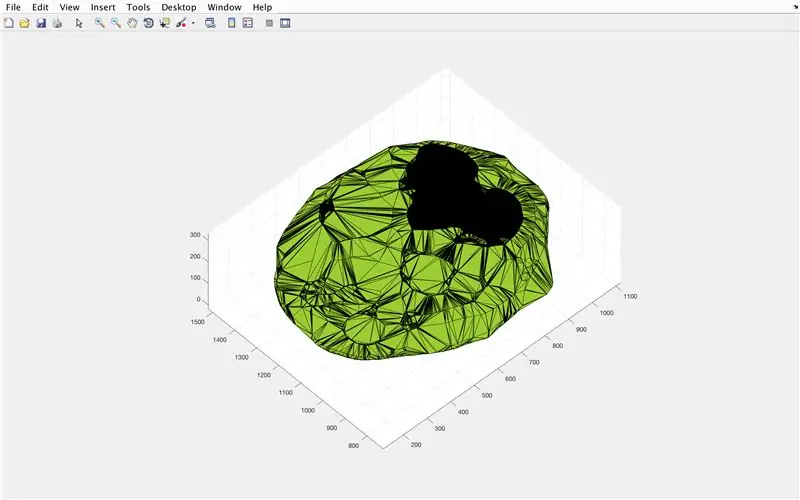
با استفاده از تابع alphaShape ، ما یک ساختار سه بعدی ایجاد می کنیم که می توانیم حجم مغز را از آن محاسبه کنیم.
- از تابع alphaShape استفاده کنید ، بردارهای "xBrain" ، "yBrain" و "z" را برای مختصات x ، y و z وصل کنید و برابر "BrainPolyhedron" قرار دهید.
- پنجره شکل 3 را باز کنید
- شکل آلفا محاسبه شده "BrainPolyhedron" را در پنجره شکل نمایش دهید
- حجم شکل آلفا را با استفاده از تابع "حجم" که برای اشکال آلفا کار می کند ، محاسبه کنید
- حجم را به میلی متر^3 تبدیل کنید
- حجم جامدات را در پنجره Command چاپ کنید
- درخواست کنید نام بیمار به عنوان ورودی تنظیم شود
- تاریخ و زمان فعلی را با ساعت دریافت کنید و روی "DateandTime" تنظیم کنید
- برای ورود و ترسیم داده های محاسبه شده ، تابع "pacientFiles" را فراخوانی کنید
از اینجا ، تصویر دوم و سوم باید ارقام ظاهر شده را نشان دهد ، و تصویر چهارم آن را که باید در پنجره فرمان نمایش داده شود نشان می دهد.
توصیه شده:
ربات هوشمند DIY ردیابی ماشین ردیابی ماشین ردیابی خودرو حساس به نور: 7 مرحله

دستگاه DIY Robot Smart Tracking Car Tracking Car Photosensitive: طراحی شده توسط SINONING ROBOT شما می توانید از ربات ردیابی ماشین خرید کنید بچرخید تا
Time Cube - ابزار ردیابی زمان آردوینو: 6 مرحله

Time Cube - ابزار ردیابی زمان آردوینو: من می خواهم یک پروژه آردوینو ساده اما واقعاً مفید را برای ردیابی رویدادهای زمان با چرخاندن برخی از ابزارهای مکعب هوشمند به شما پیشنهاد کنم. آن را به & quot؛ کار کنید & quot؛ & gt؛ & quot؛ یاد بگیر & quot؛ & gt؛ & quot؛ مشاغل & quot؛ & gt؛ & quot؛ استراحت & quot؛ طرف و آن را شمارش می کند
ردیابی GPS در زمان واقعی: 3 مرحله
ردیابی GPS در زمان واقعی: در این آموزش ، نحوه انجام ردیابی مکان در زمان واقعی با استفاده از BerryGPS-GSM ، Raspberry Pi Zero و Initial State را به شما نشان خواهیم داد. طول و عرض و عرض جغرافیایی و سرعت را از طریق 3G با BerryGPS-GSM به حالت اولیه ارسال می کنیم
لغزنده زمان گذر زمان آردوینو: 3 مرحله (همراه با تصاویر)

لغزنده زمان گذر زمان آردوینو: بنابراین من به دنبال ساختن یک ویدیوی timelapse با dslr خود بودم و بسیاری را با استفاده از مکانیزم کشویی برای افزودن بعد دیگر مشاهده کرده بودم. من به خرید یکی نگاه کردم اما خرید آنها فقط برای "فرو بردن انگشتان پا در آب" کمی گران است. بنابراین t
جعبه باربی: یک جعبه استتار/ جعبه مخصوص پخش کننده Mp3 شما: 4 مرحله (همراه با تصاویر)

جعبه باربی: یک جعبه استتار/ جعبه مخصوص پخش کننده Mp3 شما: این یک کیف محافظ محافظ برای پخش کننده mp3 شما است که جک هدفون را به یک چهارم اینچ نیز تبدیل می کند ، می تواند به عنوان یک جعبه تقویت کننده در تلنگر سوئیچ عمل کند و پخش کننده mp3 شما را به عنوان دستگاه پخش نوار اوایل دهه نود یا سرقت کم مشابه تبدیل می کند
