فهرست مطالب:
- مرحله 1: مرحله 1: بارگیری تصویر
- مرحله 2: مرحله 2: فیلتر نویز و هیستوگرام
- مرحله 3: مرحله 3: تنظیم آستانه ها
- مرحله 4: ایجاد GUI
- مرحله 5: نسخه ی نمایشی ویدئو
تصویری: تقسیم بندی ریه MatLab: 5 مرحله

2024 نویسنده: John Day | [email protected]. آخرین اصلاح شده: 2024-01-30 08:52
نویسنده: پوک لام ، پل یونگ ، اریک ریس
پذیرفتن اینکه اشتباهات در تقسیم بندی ریه ها اطلاعات نادرستی در مورد شناسایی ناحیه بیماری ایجاد می کند و می تواند مستقیماً بر روند تشخیص تأثیر بگذارد. هنگامی که بیماری های ریوی دارای اشکال چالش برانگیزی هستند ، تکنیک های مدرن کمک رایانه ای نتوانست نتایج دقیقی را ارائه دهد. این اشکال غیرطبیعی می تواند ناشی از افیوژن پلور ، ادغام و غیره باشد. با استفاده از تکنیک تقسیم بندی ریه ، که در آن مرزهای ریه از بافت اطراف قفسه سینه جدا شده است ، برنامه ما می تواند مرزها را با آستانه ورودی کاربر مشخص کند تا نمای کاملاً قابل تنظیم ایجاد کند. از شکل ریه ها ،
هدف از این پروژه MatLab ایجاد یک برنامه تقسیم بندی ریه تعاملی کاربر پسند برای تشخیص شرایط پاتولوژیک تصاویر اشعه ایکس ریه ها است. هدف ما ایجاد روش م effectiveثرتری برای نشان دادن و شناسایی ریه های غیرطبیعی است تا به پزشکان و رادیولوژیست ها روش مطمئن تری برای تشخیص بیماری های ریه ارائه شود. با استفاده از ابزار طراح برنامه در MatLab ، این برنامه به گونه ای طراحی شده است که به طور خاص با اشعه ایکس قفسه سینه و اسکن توموگرافی کامپیوتری (CT) کار می کند ، اما برای کار با اسکن MRI نیز آزمایش شده است.
دستورالعمل های زیر شامل تکنیک فیلتر نویز (فیلتر وینر کم گذر) و همچنین آستانه تصویر (با استفاده از هیستوگرام شدت تصویر خاکستری) و با استفاده از یک گرادیان مورفولوژیکی (تفاوت بین اتساع و فرسایش تصویر) تا منطقه مورد نظر را مشخص کنید سپس دستورالعمل نحوه ادغام همه عناصر در رابط کاربری گرافیکی (GUI) توضیح داده می شود.
توجه داشته باشید:
1) این پروژه از مقاله ای الهام گرفته شده است: "تقسیم بندی و تجزیه و تحلیل ریه های غیر عادی در CT: رویکردهای فعلی ، چالش ها و روندهای آینده". که در اینجا یافت می شود
2) ما از تصاویر اشعه ایکس از مرکز بالینی NIH استفاده می کنیم. پیوند را می توانید در اینجا پیدا کنید
3) راهنمای طراح برنامه را می توانید در اینجا پیدا کنید
4) قبل از اجرای کد: باید مسیر Dir (در خط 34) را به فهرست پرونده و نوع تصویر خود (خط 35) (ما در حال تجزیه و تحلیل *.png) هستیم تغییر دهید.
مرحله 1: مرحله 1: بارگیری تصویر
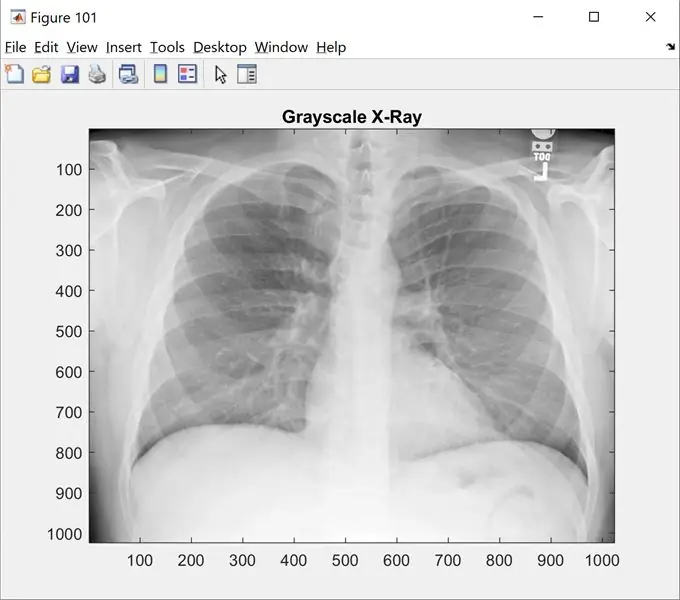
این مرحله تصویر اصلی را در مقیاس خاکستری به شما نشان می دهد. "name_of_picture.png" را به نام تصویر خود تغییر دهید
روشن clc؛ همه را بستن ؛
٪٪ بارگیری تصاویر
raw_x_ray = 'name_of_picture.png'؛
I = imread (raw_x_ray) ؛
شکل (101) ؛
نمایش (I) ؛
نقشه رنگ (خاکستری) ؛
عنوان ('اشعه ایکس مقیاس خاکستری')؛
مرحله 2: مرحله 2: فیلتر نویز و هیستوگرام
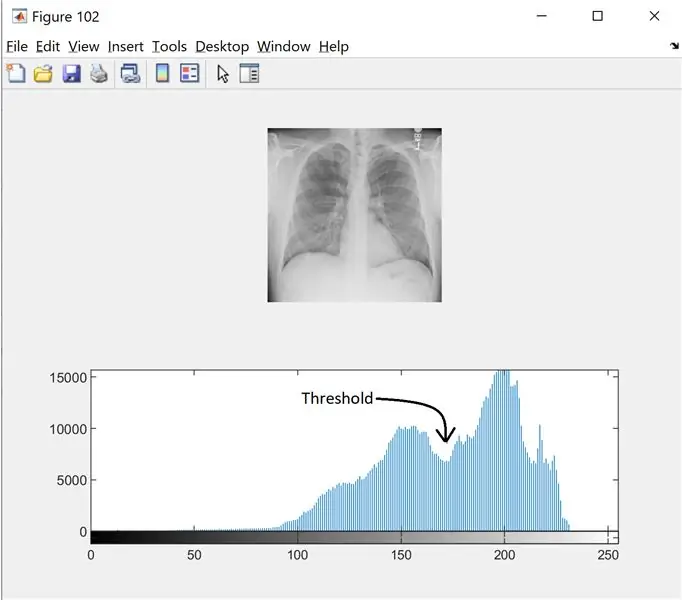
به منظور یافتن آستانه برای تصویر مقیاس خاکستری ، ما به هیستوگرام نگاه می کنیم تا ببینیم آیا حالت های متمایزی وجود دارد یا خیر. بیشتر اینجا بخوانید
I = wiener2 (من ، [5 5]) ؛
شکل (102) ؛
فرع فرعی (2 ، 1 ، 1) ؛
نمایش (I) ؛
فرع فرعی (2 ، 1 ، 2) ؛
imhist (I، 256)؛
مرحله 3: مرحله 3: تنظیم آستانه ها
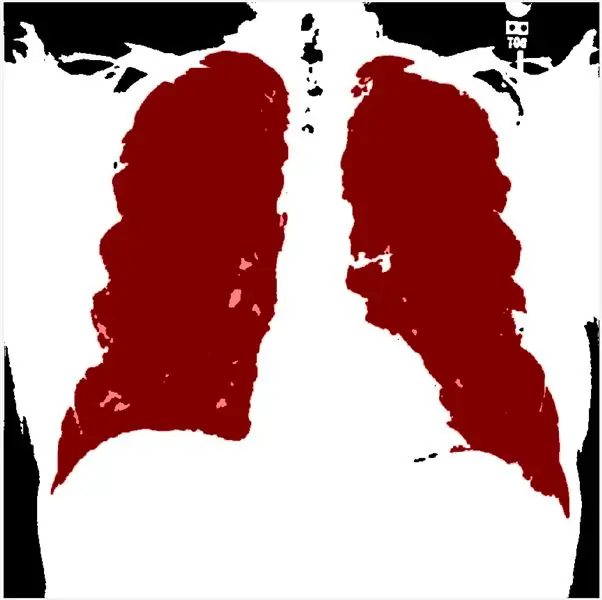
این مرحله به شما امکان می دهد تا آستانه را با توجه به هیستوگرام تنظیم کنید. morphologicalGradient ناحیه مورد نظر را با رنگ قرمز برجسته می کند و عملکرد visboundaries تصویر مشخص شده و فیلتر شده از ریه را به رنگ قرمز پوشش می دهد.
با استفاده از مناطق منطقه ای ، می توانیم آرایه های جامدت را دقیق کرده و آنها را به صورت نزولی مرتب کنیم. در مرحله بعد تصویر خاکستری sclae را باینار می کنم و روش گرادیان مورفولوژیکی و mLoren Shurasking را برای برجسته کردن منطقه مورد نظر (ROI) اعمال می کنم. گام بعدی این است که تصویر را وارونه کنید تا ROI ریه در پس زمینه سیاه سفید شود. من از تابع showMaskAsOverlay برای نمایش 2 ماسک استفاده می کنم. توجه: کد از پیوند Loren Shure الهام گرفته شده است.
به تازگی ، من با استفاده از bwbwboundaries و پوشاندن تصویر فیلتر و مرزها ، طرح قرمز ایجاد می کنم.
a_thresh = I> = 172؛ ٪ این آستانه را تعیین کرد
[labelImage، numberOfBlobs] = bwlabel (a_thresh)؛
props = regionprops (a_thresh ، "همه") ؛
sortedSolidity = مرتب سازی ([props. Solidity] ، 'فرود') ؛
SB = sortedSolidity (1) ؛
اگر SB == 1٪ SB فقط استحکام را قبول کند == 1 استخوان را فیلتر کنید
binaryImage = imbinarize (I)؛ شکل (103) ؛
imshow (binaryImage) ؛ نقشه رنگ (خاکستری) ؛
SE = strel ('مربع' ، 3) ؛
morphologicalGradient = imsubtract (imdilate (binaryImage، SE) ، imerode (binaryImage، SE)) ؛
mask = imbinarize (morphologicalGradient، 0.03)؛
SE = strel ('مربع' ، 2) ؛
mask = imclose (mask، SE)؛
mask = imfill (mask، 'hole')؛
mask = bwareafilt (mask، 2)؛ ٪ کنترل تعداد نمایش منطقه
notMask = ~ ماسک ؛
ماسک = ماسک | bwpropfilt (notMask، 'Area'، [-Inf، 5000 - eps (5000)])؛
showMaskAsOverlay (0.5 ، ماسک ، 'r') ؛ ٪ باید برنامه/عملکرد showMaskAsOverlay را بارگیری کنید
BW2 = imfill (binaryImage ، 'hole') ؛
new_image = BW2؛
new_image (~ mask) = 0 ؛ background پس زمینه و حفره های معکوس
B = bwboundaries (new_image) ؛ ٪ فقط می تواند 2 بعد را بپذیرد
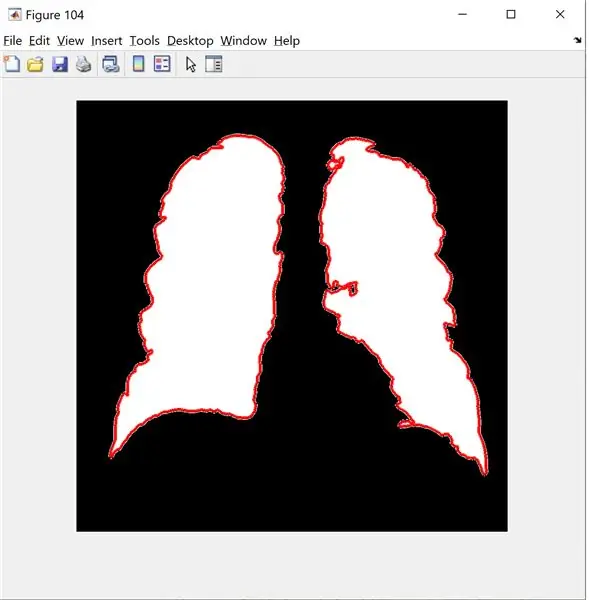
شکل (104) ؛
نمایش (new_image) ؛
صبر کن
visboundaries (B) ؛
پایان
مرحله 4: ایجاد GUI
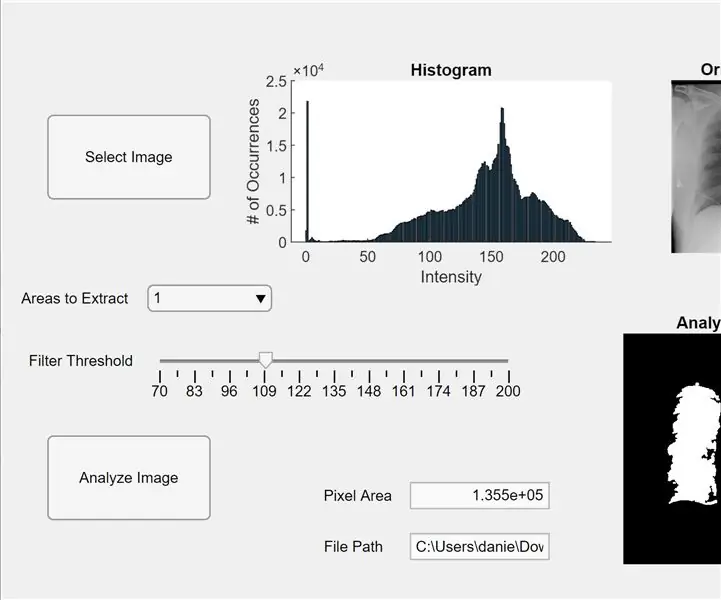
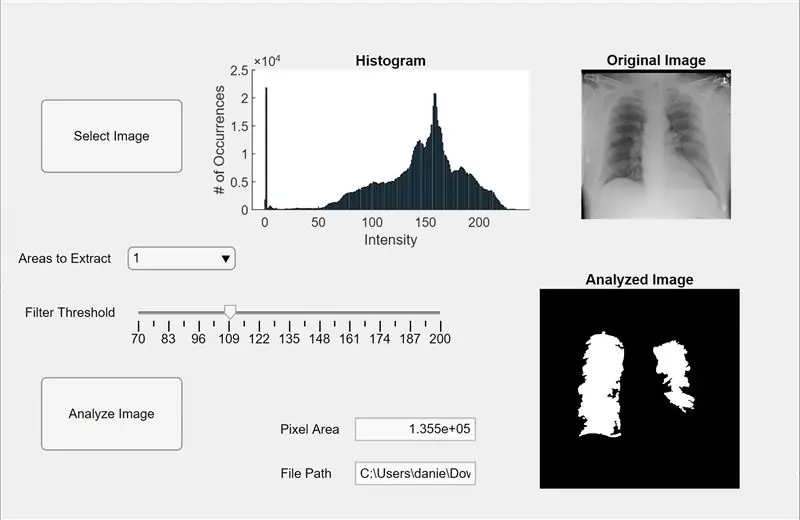
اکنون ، ما کد قبلی را در یک برنامه MATLAB ادغام می کنیم. برنامه طراح را در MATLAB (جدید> برنامه) باز کنید. ابتدا ، رابط کاربری را با کلیک-نگه داشتن و کشیدن در سه محور به فضای کار مرکزی طراحی می کنیم. در مرحله بعد ، دو دکمه ، یک زمینه ویرایش (متن) ، یک قسمت ویرایش (عددی) ، یک نوار لغزنده و یک منوی کشویی را کلیک کرده و نگه دارید. دو محور هر کدام پیش نمایش و تصویر را تجزیه و تحلیل می کنند و محورهای سوم هیستوگرام پیکسل ها را برای پیش نمایش تصویر "انتخاب شده" نمایش می دهند. کادر زمینه ویرایش (متن) مسیر فایل تصویر انتخاب شده و فیلد ویرایش (عددی) ناحیه پیکسل شناسایی شده ریه ها را نمایش می دهد.
اکنون از نمای طراحی به نمای کد در App Designer تغییر دهید. با کلیک روی دکمه قرمز "Properties" با علامت بعلاوه ، کد ویژگیها را در کد وارد کنید. ابتدا خصوصیات I ، threshold و regionToExtract را مانند کد ارائه شده در زیر تنظیم کنید. در مرحله بعد ، روی دکمه ای در بالا سمت راست محیط کار (مرورگر کامپوننت) راست کلیک کرده و از Callbacks> Go to … callback (برو به تماس) بروید. کد "function SelectImageButtonPushed (برنامه ، رویداد)" را اضافه کنید. این کد به شما امکان می دهد تا با استفاده از uigetfile تصویری را برای تجزیه و تحلیل از رایانه خود انتخاب کنید. پس از انتخاب تصویر ، یک تصویر پیش نمایش در زیر محورها همراه با هیستوگرام ظاهر می شود. سپس ، روی دکمه دیگر راست کلیک کرده و همان روش را تکرار کنید تا یک تابع callback ایجاد شود.
کد زیر را "function AnalyzeImageButtonPushed (برنامه ، رویداد)" اضافه کنید. این کد شمارش پیکسل و تشخیص لکه روی تصویر پیش نمایش را بر روی دکمه تجزیه و تحلیل تصویر (هر کدام که برای این کد راست کلیک کرده اید) انجام می دهد. پس از برنامه نویسی دکمه ها ، اکنون نوار لغزنده و منوی کشویی را برنامه ریزی می کنیم. بر روی نوار لغزنده راست کلیک کرده ، یک تابع callback ایجاد کرده و کد زیر "function FilterThresholdSliderValueChanged (برنامه ، رویداد)" را تا انتها اضافه کنید. این به نوار لغزنده اجازه می دهد تا آستانه شدت خاکستری را تنظیم کند.
یک تابع تماس برای منوی کشویی ایجاد کرده و کد زیر "تابع AreastoExtractDropDownValueChanged (برنامه ، رویداد)" را اضافه کنید تا به منوی کشویی اجازه دهید تعداد لکه های نمایش داده شده در محورهای تصویر تجزیه و تحلیل شده را تغییر دهد. اکنون ، روی هر موجود در مرورگر کامپوننت کلیک کنید و ویژگی های آنها را به دلخواه تغییر دهید ، مانند تغییر نام موجودیت ها ، حذف محورها و تغییر مقیاس. موجودات مرورگر کامپوننت را در نمای طراحی بکشید و رها کنید تا یک طرح کاربردی و آسان برای درک داشته باشید. شما اکنون یک برنامه در MATLAB دارید که می تواند تصاویر ریه ها را برای ناحیه پیکسل تجزیه و تحلیل کند!
خواص (دسترسی = خصوصی) I = ؛ ٪ فایل تصویری
آستانه = 257 ؛ ٪ آستانه برای دوتایی شدن شدت خاکستری
RegionToExtract = 2 ؛
پایان
عملکرد SelectImageButtonPushed (برنامه ، رویداد)
clc؛ Dir = 'C: / Users / danie / Downloads / images_004 / images'؛ ٪ define invariate file "prefix"
[imageExt، path] = uigetfile ('*. png')؛ ٪ قسمت متغیر نام تصویر را بگیرید
imageName = [Dir filesep imageExt]؛ ٪ رشته های متغیر متغیر و متغیر را به هم متصل می کند
app. I = imread (imageName) ؛ ٪ تصویر را خوانده است
imshow (app. I ، 'parent'، app. UIAxes) ؛ ٪ نمایش تصویر
app. FilePathEditField. Value = path؛ ٪ نمایش مسیر فایل از جایی که تصویر اصلی از آنجا آمده است
پایان
عملکرد AnalyzeImageButtonPushed (برنامه ، رویداد)
originalImage = app. I؛
originalImage = wiener2 (برنامه I. ، [5 5]) ؛ ٪ فیلتر حذف نقطه
هیستوگرام (app. AxesHistogram ، app. I، 256) ؛ ٪ نمایش هیستوگرام تصویر
a_thresh = originalImage> = app.threshold؛ ٪ این آستانه را تعیین کرد
labelImage = bwlabel (a_thresh)؛
props = regionprops (a_thresh ، 'all')؛
sortedSolidity = مرتب سازی ([props. Solidity] ، 'فرود') ؛
SB = sortedSolidity (1) ؛
اگر SB == 1٪ SB فقط استحکام را قبول کند == 1 استخوان را فیلتر کنید
SE = strel ('مربع' ، 3) ؛
morphologicalGradient = imsubtract (imdilate (labelImage، SE) ، imerode (labelImage، SE)) ؛
mask = imbinarize (morphologicalGradient، 0.03)؛
SE = strel ('مربع' ، 2) ؛
mask = imclose (mask، SE)؛
mask = imfill (mask، 'hole')؛
mask = bwareafilt (mask، app.regionsToExtract) ؛
٪ کنترل تعداد نمایش منطقه
notMask = ~ ماسک ؛
ماسک = ماسک | bwpropfilt (notMask، 'Area'، [-Inf، 5000 - eps (5000)])؛
BW2 = imfill (labelImage ، 'hole') ؛
new_image = BW2؛
new_image (~ mask) = 0 ؛
B = bwboundaries (new_image) ؛ ٪ فقط می تواند 2 بعد را نمایش دهد (new_image ، 'parent'، app. UIAxes2) ؛
نگه دارید (app. UIAxes2 ، 'on') ؛
visboundaries (B) ؛
مجموعه (gca ، 'YDir' ، 'معکوس') ؛
lungArea = bwarea (new_image) ؛
app. PixelAreaEditField. Value = lungArea؛
پایان
پایان
تابع FilterThresholdSliderValueChanged (برنامه ، رویداد)
app.threshold = app. FilterThresholdSlider. Value؛
پایان
تابع AreastoExtractDropDownValueChanged (برنامه ، رویداد) stringNumber = app. AreastoExtractDropDown. Value؛
app.regionsToExtract = str2double (stringNumber) ؛
پایان
پایان
توصیه شده:
نحوه تقسیم اعشار- EDP 279: 5 مرحله
نحوه تقسیم اعشار- EDP 279: نحوه تقسیم اعداد با اعشار
تقسیم و تمدید Lightstrip Hue Philips: 8 مرحله (همراه با تصاویر)

تقسیم و گسترش Philips Hue Lightstrip: من بیشتر & quot؛ خانه هوشمند & quot؛ را اضافه کرده ام. گجت هایی را برای خانه من تایپ کنید ، و یکی از چیزهایی که من با آن بازی می کنم Philips Hue Lightstrip است. این یک نوار چراغ LED است که می تواند از طریق یک برنامه یا از دستیار هوشمند مانند Alexa یا… کنترل شود
تقسیم کننده چیدمان Equidistant: 11 مرحله (همراه با تصاویر)

Equidistant Layout Divider: ممکن است تخصصی باشد ، اما تقسیم بندی چارچوبی ابزاری است که ممکن است به محض دستیابی به آن یک ابزار دستی را پیدا کنید. مواقعی در مغازه وجود دارد که من طول دلخواهی برای یک قطعه دارم و باید تفاوت را تقسیم کنم. به جای اندازه گیری t
فهرست بندی قالب بندی خودکار آردوینو: 3 مرحله
لیست بندی قالب بندی خودکار آردوینو: روش پیش فرض محیط برنامه نویسی آردوینو براکت ها (براکت های پیچ دار) سالهاست که من را آزار می دهد (تصویر اول را ببینید). من ترجیح می دهم براکت ها روی خطوط خودشان جدا شوند (تصویر دوم را ببینید) به من این را خیلی راحت تر می بینم
نحوه ساخت یک فیلم با صفحه تقسیم شده با چهار مرحله: 4 مرحله (همراه با تصاویر)

چگونه می توان با چهار مرحله ویدئویی با صفحه نمایش تقسیم کرد: ما اغلب می بینیم که یک نفر در صحنه ای دو بار در یک نمایش تلویزیونی نشان می دهد. و تا آنجا که ما می دانیم ، این بازیگر برادر دوقلو ندارد. ما همچنین مشاهده کرده ایم که دو ویدئوی آواز بر روی یک صفحه نمایش داده می شود تا مهارت های خوانندگی آنها مقایسه شود. این قدرت spl است
