فهرست مطالب:
- مرحله 1: یک سطل ذخیره سازی ابری Google را تنظیم کنید
- مرحله 2: داده های خود را فرمت کرده و مجموعه داده Csv ایجاد کنید
- مرحله 3: طیف سنجی های خود را در سطل خود بارگذاری کنید
- مرحله 4: Csv مجموعه داده خود را بارگذاری کنید
- مرحله 5: ایجاد مجموعه داده
- مرحله 6: مدل AutoML خود را ایجاد کنید
- مرحله 7: مدل خود را آزمایش کنید
- مرحله 8: مدل خود را در ThinkBioT نصب کنید

تصویری: قسمت 2. مدل ThinkBioT با Google AutoML: 8 مرحله

2024 نویسنده: John Day | [email protected]. آخرین اصلاح شده: 2024-01-30 08:54

ThinkBioT به عنوان "Plug and Play" با مدلهای TensorFlow Lite سازگار با Edge TPU طراحی شده است.
در این اسناد ما ایجاد طیف نگاری ، قالب بندی داده های شما و استفاده از Google AutoML را پوشش خواهیم داد.
کد در این آموزش به صورت bash نوشته می شود بنابراین با چند پلتفرم سازگار خواهد بود.
وابستگی ها
- با این حال ، قبل از شروع ، باید Sox را یک برنامه صوتی خط فرمان سازگار با دستگاه های Windows ، Mac و Linux نصب کنید.
- اگر از دستگاه Windows استفاده می کنید ، ساده ترین راه برای اجرای اسکریپت های bash استفاده از Git است ، بنابراین من توصیه می کنم بارگیری و نصب آن را از بسیاری جهات مفید بدانید ،
- برای ویرایش کد یا از ویرایشگر مورد علاقه خود استفاده کنید یا NotePad ++ را برای Windows یا Atom را برای سایر سیستم عامل ها نصب کنید.
** اگر مدل TensorFlow موجود دارید یا می خواهید آموزش را با یک مدل موجود انتقال دهید ، لطفاً به اسناد مرجانی Google مراجعه کنید.
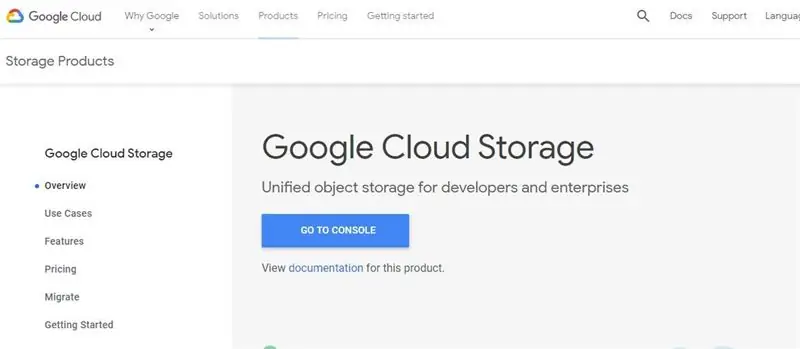
مرحله 1: یک سطل ذخیره سازی ابری Google را تنظیم کنید
1. وارد حساب جیمیل خود شوید (یا اگر حساب Google ندارید یک حساب ایجاد کنید)
2. به صفحه انتخاب پروژه بروید و یک پروژه جدید برای فایل های مدل و طیف نگاری خود بسازید. برای پیشرفت بیشتر باید صورت حساب را فعال کنید.
3. از https://cloud.google.com/storage/ دیدن کنید و دکمه ایجاد سطل را در بالای صفحه فشار دهید.
4. نام سطل مورد نظر خود را وارد کرده و سطل را با قبول تنظیمات پیش فرض ایجاد کنید.
مرحله 2: داده های خود را فرمت کرده و مجموعه داده Csv ایجاد کنید
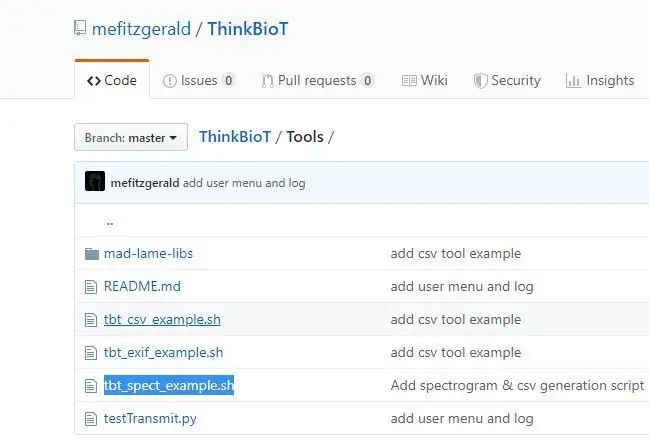
من یک اسکریپت مفید برای ایجاد فایل dataset.csv شما که برای ایجاد مدل مورد نیاز است طراحی کرده ام. فایل مجموعه داده ها تصاویر موجود در سطل شما را به برچسب های موجود در مجموعه داده پیوند می دهد.
1. مخزن ThinkBioT را از GitHub بارگیری کنید و
2. فایل tbt_spect_example.sh را از فهرست ابزارها در یک پوشه جدید روی دسکتاپ خود کپی کنید.
3. فایلهای صوتی موردنظر خود را در مدل خود اضافه کنید و آنها را در پوشه هایی با برچسب خود قرار دهید (به عنوان مثال آنچه را که می خواهید مرتب شوند. به عنوان مثال ، اگر می خواهید سگ یا گربه را شناسایی کنید ، می توانید یک پوشه داشته باشید سگ ، با صداهای پوست یا پوشه ای به نام گربه با صداهای گربه و غیره.
4. tbt_spect_example.sh را با Notepad ++ باز کنید و "yourbucknamenename" در خط 54 را با نام سطل ذخیره سازی Google خود جایگزین کنید. به عنوان مثال ، اگر سطل شما myModelBucket نامیده می شد ، خط به
سطل = "gs: // myModelBucket/spectro-data/"
5. کد را با تایپ موارد زیر در ترمینال Bash خود اجرا کنید ، کد اجرا می شود و فایل csv برچسب ها و یک فهرست به نام spectro-data در بالای میز شما با طیف نگاری های ایجاد شده ایجاد می شود.
sh tbt_spect_example.sh
مرحله 3: طیف سنجی های خود را در سطل خود بارگذاری کنید
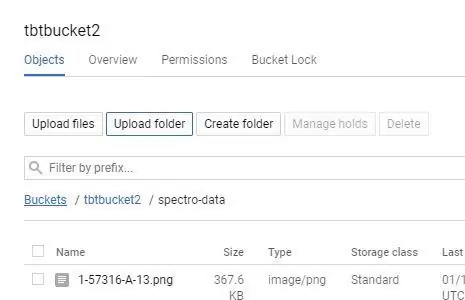
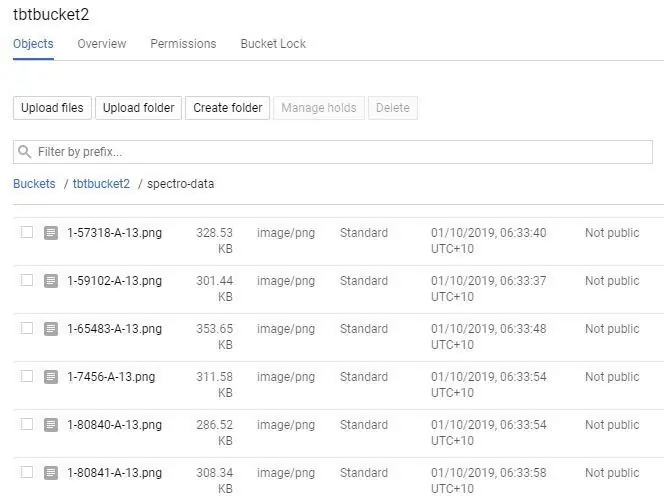
چند روش برای بارگذاری در Google Storage وجود دارد ، ساده ترین راه این است که یک پوشه مستقیم را بارگذاری کنید.
1. روی نام سطل خود در صفحه Google Storage خود کلیک کنید.
2. دکمه "UPLOAD FOLDER" را انتخاب کرده و فهرست "spectro-data/" خود را که در آخرین مرحله ایجاد شده است انتخاب کنید.
یا
2. اگر تعداد زیادی فایل دارید ، می توانید با انتخاب "ایجاد پوشه" ، فهرست "spectro-data/" را به صورت دستی ایجاد کنید ، سپس وارد پوشه شده و "UPLOAD FILES" را انتخاب کنید. این می تواند یک گزینه عالی برای مجموعه داده های بزرگ باشد زیرا می توانید طیف نگاری ها را در بخش ها بارگذاری کنید ، حتی با استفاده از چندین رایانه برای افزایش سرعت بارگذاری.
یا
2. اگر کاربر پیشرفته ای هستید می توانید از طریق Google Cloud Shell بارگذاری کنید.
gsutil cp spectro-data/* gs: // your-bucket-name/spectro-data/
اکنون باید سطل پر از طیف نگارهای بسیار زیبا داشته باشید!
مرحله 4: Csv مجموعه داده خود را بارگذاری کنید
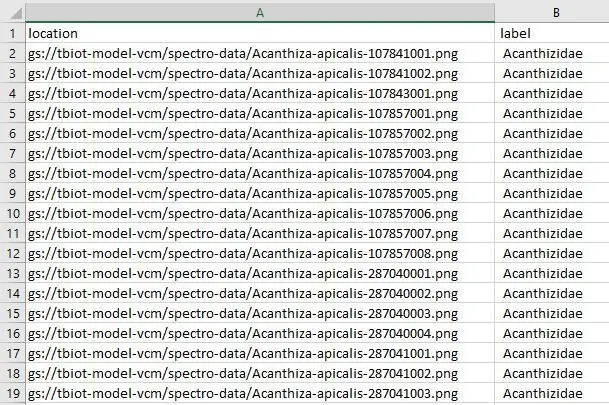
اکنون ما باید فایل model-labels.csv را در فهرست "spectro-data/" شما در Google Storage بارگذاری کنیم ، اساساً همان مرحله آخر است ، شما فقط یک فایل را به جای تعداد زیادی بارگذاری می کنید.
1. روی نام سطل خود در صفحه Google Storage خود کلیک کنید.
2. دکمه UPLOAD FILE را انتخاب کرده و فایل model-labels.csv خود را که قبلاً ایجاد کرده اید انتخاب کنید.
مرحله 5: ایجاد مجموعه داده
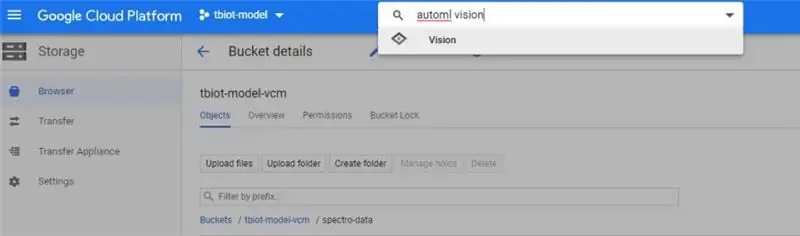
1. ابتدا باید API AutoML VIsion را پیدا کنید ، ممکن است کمی مشکل باشد! ساده ترین راه این است که "automl vision" را در نوار جستجوی ذخیره سازی Google Cloud خود (در تصویر) جستجو کنید.
2. هنگامی که روی پیوند API کلیک می کنید ، باید API را فعال کنید.
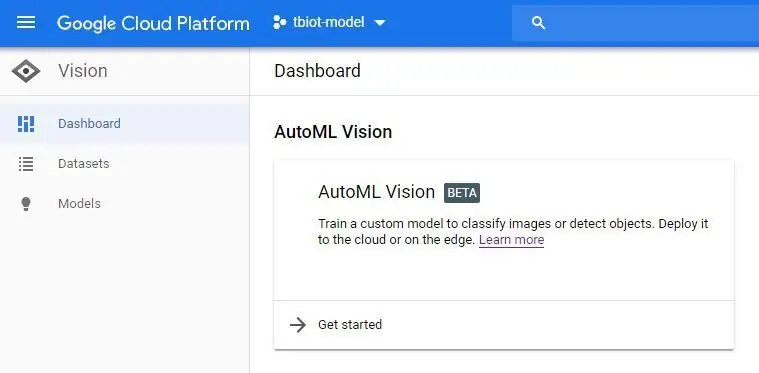
3. اکنون در داشبورد AutoML Vision (تصویر) روی دکمه مجموعه داده جدید کلیک کرده و Single label و گزینه "Select a CSV file" را انتخاب کنید. سپس پیوند فایل model-labels.csv خود را در سطل ذخیره سازی خود قرار می دهید. اگر این آموزش را دنبال کرده اید مطابق شکل زیر خواهد بود
gs: //yourBucketName/spectro-data/model-labelsBal.csv
4. سپس ادامه را فشار دهید تا مجموعه داده شما ایجاد شود. ایجاد آن ممکن است مدتی طول بکشد.
مرحله 6: مدل AutoML خود را ایجاد کنید
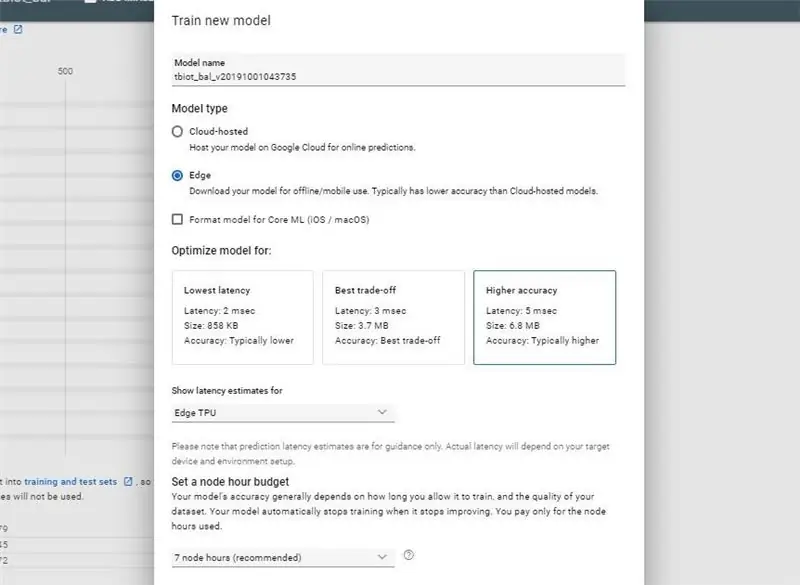

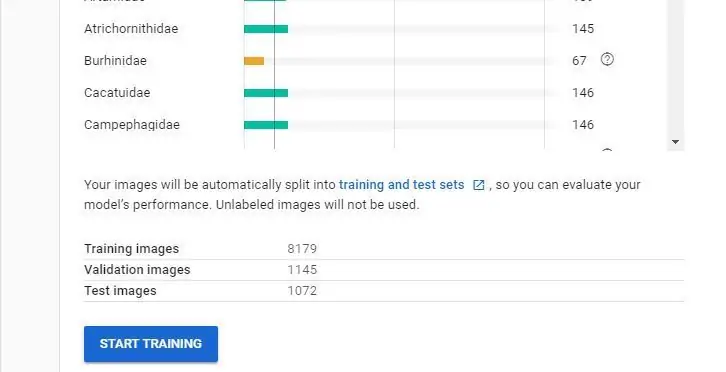
هنگامی که ایمیل خود را دریافت کردید که به شما اطلاع می دهد مجموعه داده شما ایجاد شده است ، آماده ایجاد مدل جدید خود هستید.
- دکمه TRAIN را فشار دهید
- نوع مدل را انتخاب کنید: برآورد تاخیر لبه و مدل: Edge TPU و سایر گزینه ها را به صورت پیش فرض در ابتدا بگذارید ، ممکن است بعداً آزمایش کنید.
- اکنون مدل شما آموزش می بیند ، مدتی طول می کشد و هنگامی که آماده بارگیری شد ، ایمیلی دریافت خواهید کرد.
توجه: اگر دکمه آموزش در دسترس نیست ممکن است با مجموعه داده خود مشکل داشته باشید. اگر کمتر از 10 کلاس (برچسب) دارید ، سیستم به شما اجازه آموزش مدل نمی دهد ، بنابراین ممکن است مجبور شوید تصاویر بیشتری اضافه کنید. اگر نیاز به توضیح دارید ، باید به Google AutoML Video نگاهی بیندازید.
مرحله 7: مدل خود را آزمایش کنید
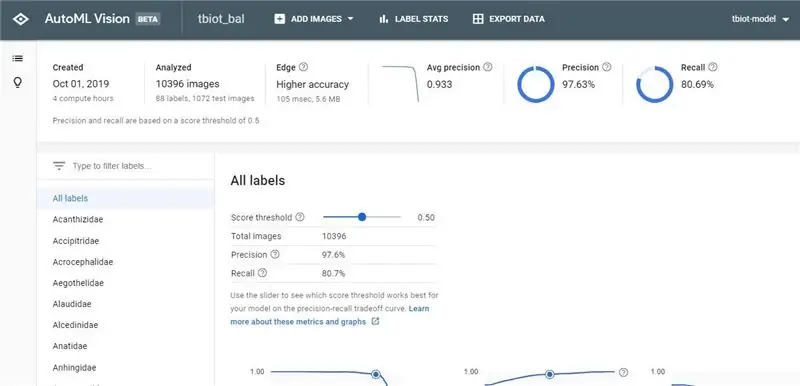
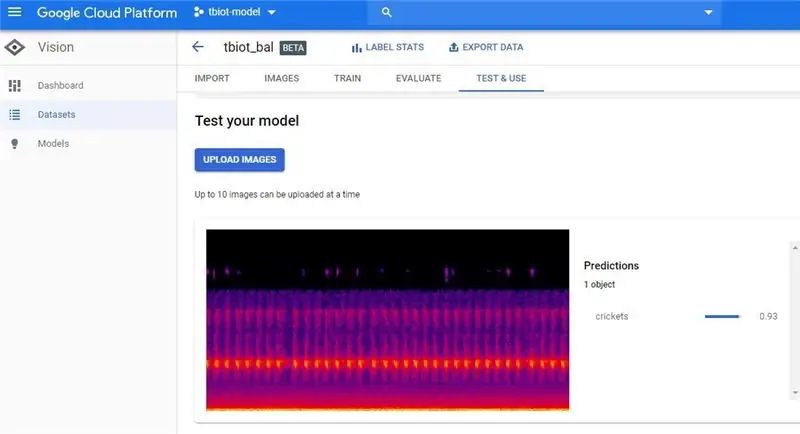
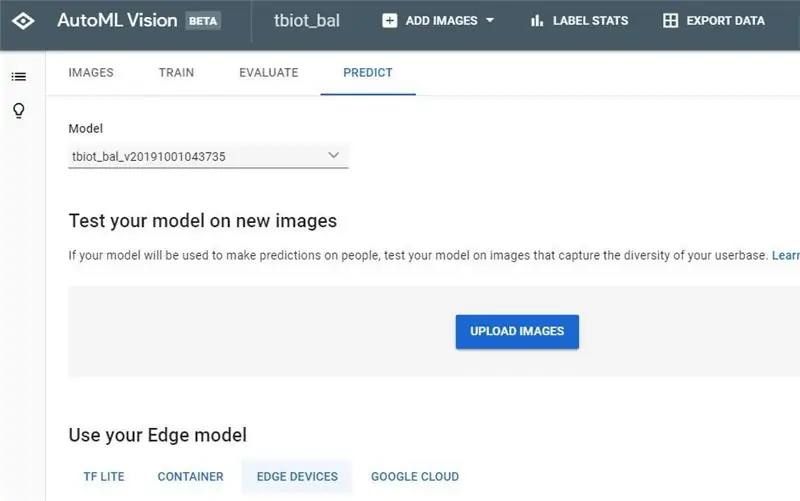
پس از دریافت ایمیل تکمیل مدل خود ، روی پیوند کلیک کنید تا به API AutoML Vision بازگردید.
1. اکنون می توانید نتایج و ماتریس گیجی مدل خود را مشاهده کنید.
2. گام بعدی این است که مدل خود را آزمایش کنید ، به "TEST & USE" یا "PREDICT" بروید ، عجیب است که به نظر می رسد 2 رابط کاربری گرافیکی کاربر وجود دارد که هر دو آنها را تصویر کرده ام ، اما گزینه های هر دو دارای عملکرد یکسانی هستند.
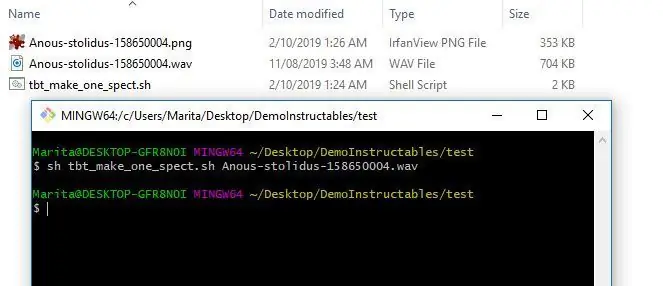
3. اکنون می توانید طیف نگاری آزمایشی را بارگذاری کنید. برای ایجاد یک طیف نگار واحد می توانید از برنامه tbt_make_one_spect.sh از ThinkBioT Github استفاده کنید. کافی است آن را در پوشه ای با wav که می خواهید به طیف نویسی تبدیل کنید ، یک پنجره Git Bash (یا ترمینال) باز کنید و از کد زیر استفاده کنید و نام فایل خود را جایگزین کنید.
sh tbt_make_one_spect.sh yourWavName.wav
4. اکنون به سادگی طیف نگار را بارگذاری کرده و نتیجه خود را بررسی کنید!
مرحله 8: مدل خود را در ThinkBioT نصب کنید
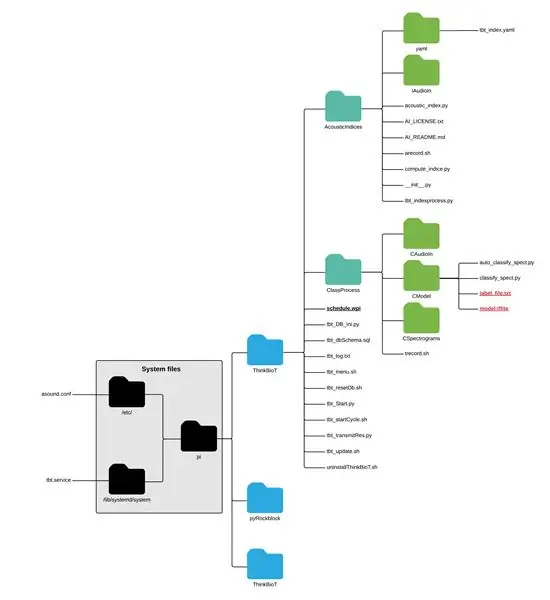
برای استفاده از مدل براق جدید خود کافی است مدل و فایل txt را در پوشه CModel رها کنید.
pi> ThinkBioT> ClassProcess> CModel
اکنون شما آماده استفاده از ThinkBioT هستید:)
** NB ** اگر از مدل خود در خارج از چارچوب ThinkBioT استفاده می کنید ، باید سند برچسب خود را به عنوان اعداد اضافه در ابتدای هر خط ویرایش کنید ، زیرا جدیدترین مفسرهای tflite ساخته شده در تابع "readlabels" آنها را فرض می کند. من یک تابع سفارشی در چارچوب ThinkBioT classify_spect.py به عنوان اثری نوشته ام که می توانید از آن در کد خود استفاده کنید:)
def ReadLabelFile (file_path):
counter = 0 with open (file_path، 'r'، encoding = 'utf-8') as f: lines = f.readlines () ret = {} for line in lines: ret [int (counter)] = line.strip () counter = counter + 1 ret ret
توصیه شده:
Miniaturizing Arduino Autonomous Robot (Land Rover / Car) Stage1 مدل 3: 6 مرحله

Miniaturizing Arduino Autonomous Robot (Land Rover / Car) Stage1Model3: تصمیم گرفتم لندرور / خودرو / ربات را کوچک کنم تا اندازه و مصرف برق پروژه کاهش یابد
مدل راه آهن مدل خودکار با حلقه های معکوس: 14 مرحله

طرح خودکار مدل راه آهن با حلقه های معکوس: در یکی از دستورالعمل های قبلی من ، نحوه ساخت یک راه آهن ساده نقطه ای به نقطه مدل ساده را نشان دادم. یکی از معایب اصلی آن پروژه این بود که قطار برای برگشت به نقطه شروع باید در جهت معکوس حرکت کند. آر
قسمت 1. ThinkBioT سنسور خودکار بیولوژیکی آکوستیک سخت افزار: 13 مرحله

بخش 1. ThinkBioT Autonomous Bio-acoustic Sensor Hardware Build: ThinkBioT با ارائه جزئیاتی از جمع آوری داده ها ، پیش پردازش ، انتقال داده ها و تجسم وظایف ، یک چارچوب نرم افزاری و سخت افزاری را طراحی می کند که به عنوان پشتوانه فناوری برای حمایت از تحقیقات بیشتر طراحی شده است. محقق
نحوه ایجاد اولین اقدام خود برای Google Home (در 10 دقیقه) قسمت 1: 10 مرحله
چگونه می توان اولین اقدام خود را برای Google Home ایجاد کرد (در 10 دقیقه) قسمت 1: سلام ، این اولین مورد از سری مقالاتی است که من در آن می نویسم و در آن نحوه توسعه و استقرار Actions در Google را یاد می گیریم. در واقع ، من روی "اقدامات در Google" چند ماه گذشته کار می کنم. من مقالات زیادی را در مورد
مدل سازی دست در مایا و گل آلود (قسمت 1): 9 مرحله
مدل سازی دست در مایا و Mudbox (قسمت 1): مایا یک برنامه Autodesk عالی است که می تواند برای یک کاربر جدید کاملاً ترسناک باشد. اگر با یک بخش از مایا مانند مدل سازی شروع کنید ، راهی عالی برای آشنایی با برنامه است. اجازه دهید با ایجاد یک قفسه سفارشی برای دسترسی سریع به چند ابزار برای شروع
